W modelowaniu molekularnym spotykamy różne formaty zapisu struktury związków, najpopularniejszymi formatami są układ kartezjański x,y,z, z-macierz, .pdb, czy .cif. Prawda jest taka, że istnieje mnóstwo formatów zapisu, metod konwersji, rodzaju przydatności czy oprogramowania.
Dlaczego tak dużo formatów zapisów struktur do modelowania molekularnego?
Tak jak w muzyce spotyka się całą gamę plików np.: ogg, mp3, mp4, acc, także i w chemii molekularnej spotyka się. Spowodowane jest to wygodą przedstawiania struktury oraz danych i tak np.: cif – zoptymalizowany jest do przechowywania informacji o strukturze krystalograficznej, a pdb do przechowywania informacji o białkach. Formaty różne są tak opracowywane, aby najbardziej odpowiadały oczekiwaniom producentów oprogramowania – optymalizacja wydajności i przechowywania danych także i tych dodatkowych
Jakie są formaty zapisów struktur do modelowania molekularnego?
Tak jak powyżej wspomnieliśmy jest cała gama formatów (.log, .inp, gjf, .acc, .png2, .gzmat, .pdb, .cube, .fchk, .chk, . Jin, .k, .mol, .mol2, .ml2 etc...)
Jak przekonwertować (zamienić jeden format zapisu na drugi format zapisu)?
Jeżeli zdarzy się problem czytania jednego formatu przez program, wówczas najlepszym sposobem jest zmiana formatu. Do tego celu można skorzystać z różnych programów, ale najlepszym rozwiązaniem jest skorzystanie z programu OPEN BABEL, który oferuję w prosty sposób konwersję na różne formaty. Po uruchomieniu tego oprogramowania widzimy dwa główne okna, w jednym oknie (prawym) wklejamy strukturę którą chcemy przekonwertować, ustawiamy bieżący format, a następnie w drugim oknie wybieramy format docelowy. I tak struktura etanolu – możemy przedstawić w postaci:
======================================
.mol2, .ml2 – Sybyl
# STRUKTURA czasteczki
# Etanol
#
#
@<TRIPOS>MOLECULE
Molecule Name
9 8
SMALL
NO_CHARGES
@<TRIPOS>ATOM
1 C1 -0.5134 0.5357 0.0000 C
2 H2 -0.1567 -0.4731 0.0000 H
3 H3 -0.1567 1.0401 -0.8737 H
4 H4 -1.5834 0.5357 0.0000 H
5 C5 -0.0001 1.2617 1.2574 C
6 H6 -0.3551 2.2710 1.2564 H
7 H7 -0.3583 0.7584 2.1311 H
8 O8 1.4299 1.2594 1.2587 O
9 H9 1.7504 1.7119 2.0424 H
@<TRIPOS>BOND
1 1 2 1
2 1 3 1
3 1 4 1
4 1 5 1
5 5 6 1
6 5 7 1
7 5 8 1
8 8 9 1
======================================
.xyz-układ kartezjański
9
Molecule Name
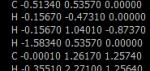
C -0.51340 0.53570 0.00000
H -0.15670 -0.47310 0.00000
H -0.15670 1.04010 -0.87370
H -1.58340 0.53570 0.00000
C -0.00010 1.26170 1.25740
H -0.35510 2.27100 1.25640
H -0.35830 0.75840 2.13110
O 1.42990 1.25940 1.25870
H 1.75040 1.71190 2.04240
======================================
.tmt – turbomole coordinate format
$coord
-0.97018539257171 1.01232628515907 0.00000000000000 c
-0.29612008378650 -0.89402942973447 0.00000000000000 h
-0.29612008378650 1.96550414260583 -1.65105371540690 h
-2.99219234631485 1.01232628515907 0.00000000000000 h
-0.00018897261250 2.38426745190441 2.37614162956694 c
-0.67104174698522 4.29156802986044 2.37425190344195 h
-0.67708887058520 1.43316829319514 4.02719534497384 h
2.70211938612833 2.37992108181693 2.37859827352943 o
3.30777660918878 3.23502215337653 3.85957663768691 h
$end
======================================
.pdb – Protein databank
COMPND Molecule Name
AUTHOR GENERATED BY OPEN BABEL 2.3.2
HETATM 1 C LIG 1 -0.513 0.536 0.000 1.00 0.00 C
HETATM 2 H LIG 1 -0.157 -0.473 0.000 1.00 0.00 H
HETATM 3 H LIG 1 -0.157 1.040 -0.874 1.00 0.00 H
HETATM 4 H LIG 1 -1.583 0.536 0.000 1.00 0.00 H
HETATM 5 C LIG 1 -0.000 1.262 1.257 1.00 0.00 C
HETATM 6 H LIG 1 -0.355 2.271 1.256 1.00 0.00 H
HETATM 7 H LIG 1 -0.358 0.758 2.131 1.00 0.00 H
HETATM 8 O LIG 1 1.430 1.259 1.259 1.00 0.00 O
HETATM 9 H LIG 1 1.750 1.712 2.042 1.00 0.00 H
CONECT 1 2 3 4 5
CONECT 1
CONECT 2 1
CONECT 3 1
CONECT 4 1
CONECT 5 1 6 7 8
CONECT 5
CONECT 6 5
CONECT 7 5
CONECT 8 5 9
CONECT 9 8
MASTER 0 0 0 0 0 0 0 0 9 0 9 0
END
======================================
.adf – ADF cartesian input format
TITLE Molecule Name
CHARGE 0 0
Number of atoms
9
ATOMS Cartesian
C -0.51340 0.53570 0.00000
H -0.15670 -0.47310 0.00000
H -0.15670 1.04010 -0.87370
H -1.58340 0.53570 0.00000
C -0.00010 1.26170 1.25740
H -0.35510 2.27100 1.25640
H -0.35830 0.75840 2.13110
O 1.42990 1.25940 1.25870
H 1.75040 1.71190 2.04240
End
Basis
End
Geometry
End
======================================
.cif – crystalografic data format
# CIF file generated by openbabel 2.3.2, see http://openbabel.sf.net
data_I
_chemical_name_common 'Molecule Name'
loop_
_atom_site_type_symbol
_atom_site_label
_atom_site_Cartn_x
_atom_site_Cartn_y
_atom_site_Cartn_z
C C1 -0.51340 0.53570 0.00000
H H2 -0.15670 -0.47310 0.00000
H H3 -0.15670 1.04010 -0.87370
H H4 -1.58340 0.53570 0.00000
C C5 -0.00010 1.26170 1.25740
H H6 -0.35510 2.27100 1.25640
H H7 -0.35830 0.75840 2.13110
O O8 1.42990 1.25940 1.25870
H H9 1.75040 1.71190 2.04240
======================================
.gamin – GAMMES format
$CONTRL COORD=CART UNITS=ANGS $END
$DATA
Molecule Name
C1
C 6.0 -0.5134000000 0.5357000000 0.0000000000
H 1.0 -0.1567000000 -0.4731000000 0.0000000000
H 1.0 -0.1567000000 1.0401000000 -0.8737000000
H 1.0 -1.5834000000 0.5357000000 0.0000000000
C 6.0 -0.0001000000 1.2617000000 1.2574000000
H 1.0 -0.3551000000 2.2710000000 1.2564000000
H 1.0 -0.3583000000 0.7584000000 2.1311000000
O 8.0 1.4299000000 1.2594000000 1.2587000000
H 1.0 1.7504000000 1.7119000000 2.0424000000
$END
======================================
.gzmat, .zmat – z-macierz dla plików Gaussiana
#Put Keywords Here, check Charge and Multiplicity.
Molecule Name
0 1
C
H 1 r2
H 1 r3 2 a3
H 1 r4 2 a4 3 d4
C 1 r5 2 a5 3 d5
H 5 r6 1 a6 2 d6
H 5 r7 1 a7 2 d7
O 5 r8 1 a8 2 d8
H 8 r9 5 a9 1 d9
Variables:
r2= 1.0700
r3= 1.0700
a3= 109.47
r4= 1.0700
a4= 109.47
d4= 120.00
r5= 1.5400
a5= 109.47
d5= 240.00
r6= 1.0699
a6= 109.47
d6= 179.88
r7= 1.0700
a7= 109.47
d7= 299.89
r8= 1.4300
a8= 109.47
d8= 59.89
r9= 0.9600
a9= 109.50
d9= 179.97
Podsumowanie formatów zapisów struktur
Jak widzimy, istnieje naprawdę wiele formatów plików, które różnią się budową pliku oraz sposobem zapisu. Te kilka przykładów struktury etanolu przekonwertowanej na różne formaty świadczą o tym. Konwersje można przeprowadzać programem openbabel lub obabel strona: openbabel.org.
==============
Import do pliku .stl - druk 3D cząsteczek
Czasami jest to użyteczne, aby dokonać konwersji plików do .stl lub .bfbe. Obiekt w postaci takiego pliku można wydrukować na większości drukarek 3D. Aby sprostać potrzebom rynku oferujemy naszym klientom konwersji plików do wymaganego formatu. Ze względu na dużą liczbę możliwości opisu struktury, podchodzimy do każdego zlecenia indywidualne, oferując najwyższą jakość usług.
Struktura cząsteczki
Typowe formaty stosowane w inforatyce chemicznej:Chemical Markup Language (cml, mrv), InChI format (inchi), InChIKey (inchikey), MDL MOL format (mol, mdl, sdf, sd), Protein Data Bank format (pdb, ent), SMILES format (smi, smiles) Sybyl Mol2 format (ml2, sy2, mol2), Formaty użytkowe porównanie cząsteczek za pomocą InChI (k), Copy raw text (copy), Fastsearch format (fs) Fingerprint format (fpt), General XML format (xml), Generic Output file format (dat, output, out, log), MolPrint2D format (mpd), Multilevel Neighborhoods of Atoms (MNA) (mna), Open Babel molecule report (molreport), Open Babel report format (report) Outputs nothing (nul), Read and write raw text (text), Title format (txt) XYZ cartesian coordinates format (xyz), Inne formaty Accelrys/MSI Biosym/Insight II CAR format (arc, car), Accelrys/MSI Cerius II MSI format (msi), Accelrys/MSI Quanta CSR format (csr), MCDL format (mcdl) MSI BGF format (bgf), PubChem format (pc), Formaty chemii obliczeniowej ABINIT Output Format (abinit), ADF cartesian input format (adf), ADF output format (adfout), CAChe MolStruct format (cache, cac), CASTEP format (castep), Cacao Cartesian format (caccrt), Cacao Internal format (cacint), DMol3 coordinates format(outmol, dmol), FHIaims XYZ format (fhiaims), Fenske-Hall Z-Matrix format (fh), GAMESS Input (inp, gamin), GAMESS Output (gamout, gam, gamess), GAMESS-UK Input (gukin), GAMESS-UK Output (gukout), GULP format (got), Gaussian 98/03 Input (gjf, gjc, gau, com), Gaussian Output (g09, g03, g94, g92, g98, gal), Gaussian Z-Matrix Input (gzmat), Gaussian formatted checkpoint file format (fchk, fch, fck), HyperChem HIN format (hin), Jaguar input format (jin), Jaguar output format (jout) MOPAC Cartesian format (mopcrt, mpc, mop), MOPAC Internal (mopin), MOPAC Output format (moo, mopout), MPQC output format (mpqc), MPQC simplified input format (mpqcin), Molpro input format (mp), Molpro output format (mpo), NWChem input format (nw), NWChem output format (nwo), PWscf format (pwscf), Parallel Quantum Solutions format (pqs), Q-Chem input format (qcin), Q-Chem output format (qcout), TurboMole Coordinate format (tmol), VASP format (CONTCAR, POSCAR), ZINDO input format (zin), Formaty krystalografii ACR format (acr), CSD CSSR format (cssr), Crystallographic Information File (cif) Free Form Fractional format (fract), Macromolecular Crystallographic Info (mmcif, mcif) ShelX format (ins, res), Reaction formats, CML Formaty reakcji (cmlr), MDL RXN format (rxn), Reaction SMILES format (rsmi), Image formats, PNG 2D depiction (png), PNG2 format (png2), POV-Ray input format (pov), SVG 2D depiction (svg), 2D drawing formats, ChemDraw CDXML format (cdxml), ChemDraw Connection Table format (ct), ChemDraw binary format (cdx), Chemical Resource Kit diagram(2D) (crk2d), Chemtool format (cht), 3D viewer formats, Ball and Stick format (bs), ADF TAPE41 format (t41), Gaussian cube format (cube, cub) itp...
Drukowanie 3D - STL Format (ASCII i Binnarny) i .bfbe
STL (STereoLitografia) to format pliku pochodzi z oprogramowania stworzonego do stereolitografii systemów CAD przez 3D Systems. STL jest również znany jako Standard Tessellation Language. Ten format pliku jest szeroko stosowany do szybkiego prototypowania i wytwarzania obiektów. Pliki STL opisują tylko geometrię powierzchni trójwymiarowego obiektu bez reprezentacji koloru, faktury lub innych cech obiektu. Format STL określa reprezentacje zarówno w ASCII i binarnie. Pliki binarne są bardziej powszechne, ponieważ są mniejszego rozmiaru. Pliki STL mają formę tekstowych poleceń w tym instrukcji wykonywanych w pętli, które określają siatkę wielokątów.
.bfbe jest formatem 3D Systems. 3D Systems jest wiodącym dostawcą druku 3D, w tym drukarek 3D.
Zastosowanie drukowania modeli 3D:
- w edukacji:
- wizualizacja cząsteczek
- modelowanie ścieżki reakcji
- modele solwatacji
- badania oddziaływań enzymów-ligandów
- modele tworzenia soli
- stereochemia
- terminologia nauka
- w laboratoriach badawczo-rozwojowych:
- instrukcja dokowania białek
- w inżynierii materiałowej - projektowanie materiałów, takich jak grafenu, fullerenu
- w procesie absorpcji
- w określaniu wielkości porów
- w konstrukcji katalizatorów
- w spintronice
- narzędziach modelowania molekularnych
- prezentacja naukowa
- w marketingu:
- materiały marketingowe
- prezentacje
- ilustracje występujących zjawisk
- modele przestrzenne
- ilustracje do czasopism i artykułów
- inne:
- biżuteria
- pamiątki
- animacja sztuki
- prezenty dla przełożonego