Praktyczna szkoła Modelowania I – 2009 - organizowana była wraz z ICM, pierwsza szkoła (22 osoby) zagadnienia z Gaussian, Hyperchema, oraz z innych programów modelowania.
Idea praktycznej Szkoły Modelowania Molekularnego narodziła się w wakacje 2009 roku. Była zainspirowana pobytem w Stanach Zjednoczonych w 2005 roku i uczestnictwem w szkoleniu organizowanym przez profesora Jerzego Leszczyńskiego, które odbyło się w Jackson State University. Zdobywanie wiedzy w Praktycznej Szkole Modelowania Molekularnego polega na rozwiązywaniu określonych problemów badawczych -rozwiązując je, uczestnik zdobywa informacje o narzędziach, a przede wszystkim o sposobie rozwiązania. Organizacja nauki takim sposobem jest trudniejsza, ale zamierzenie, jakim jest nauka analitycznego myślenia, rozwiązywania poprzez analogię, dedukcję, indukcję, abdukcję wydaje się o wiele cenniejsza niż sama wiedza z danej dziedziny. Dlatego stworzyliśmy szkołę łączącą wiedzę z dziedziny modelowania molekularnego wraz z praktycznym rozwiązywaniem problemów z dziedziny chemii, nanotechnologii, biotechnologii. Szkoła opiera się na pierwszym w Polsce serwisie internetowym do modelowania molekularnego i nie tylko: molnet.eu
Poniżej przedstawiamy dokumenty, informację już archiwalne związane z tą szkołą. Niektóre zagadnienia zostały poprawione i dołączone do kategorii szczegółowych serwisu molnet.eu
Przykład projektu badawczego na Praktyczną Szkołę Modelowania Molekularnego I
Modelowanie mechanizmu syntezy wiązania P-C
Prowadzący: Marek Doskocz
Realizacja: Karolina Gluza
Poziom trudności: zaawansowany
Modelowanie mechanizmu odwrotnego do mechanizmu rozpadu wiązania C-P opisanego w publikacji: Doskocz, M.; Majumdar, D.; Doskocz, J.; Roszak, S.; Gancarz, R.; Leszczynski, J., Theoretical studies on the mechanism of C-P bond cleavage of a model α-aminophosphonate in acidic condition. J. Phys. Chem. A 2008. 112, 2077
Cel: Nauka oraz opracowanie danych.
Projekt - Praktyczna Szkoła Modelowania Molekularnego
Hybrydyzacja w prostych związkach węglowych
Prowadzący: Piotr Wojciechowski, pomysł M. Doskocz
Poziom trudności: podstawowy
Celem projektu jest obliczenie konturów orbitali dla prostych związków węglowych: metanu, etanu, etenu, acetylenu, benzenu. Przeprowadzenie analizy NBO. Wykonanie ilustracji.
Schemat:
-
studia literaturowe na dany temat.
-
obliczenia teoretyczne (optymalizacja, Freq, NBO) prostych cząsteczek
-
interpretacja oraz wizualizacja danych
Spodziewane rezultaty: dydaktyczny, publikacja rysunków w Wikipedii, jeżeli wyniki uzyskane będą zadowalające, oraz uczestnik wyrazi chęć dalszej pracy, przygotowanie artykułu dydaktycznego na przykład do czasopisma Chemia w Szkole.
Projekt Badawczy Na Praktyczną Szkołę Modelowania Molekularnego I - przykład
Pierwszym etapem jest skopiowanie plików na maszynę dostępową Delta (gw.icm.edu.pl). Założymy, że wkopiowaliśmy katalog razem z plikami do obliczeń i nazwaliśmy go MOLL.
Następnie musimy przekopiować ten katalog na maszynę Halo, na której docelowo będziemy wykonywać obliczenia. Wykonujemy zatem polecenie:
scp -vr MOLL halo:~
(Wytłumaczenie: scp – kopiuje, vr – opcje, wyświetla informację o kopiowaniu oraz kopiuje wszystkie pliki zawarte w katalogu wraz z katalogiem)
Po wykonaniu tego polecenia musimy podać hasło, takie samo jak przy logowaniu na maszynę dostępową Dleta (gw.icm.edu.pl). Następnie logujemy się na maszynę Halo, poleceniem:
ssh halo
Po podaniu hasła, możemy sprawdzić zawartość naszego katalogu domowego poleceniem:
ls
Następnie katalogu MOLL:
ls MOLL
Teraz przystępujemy do przygotowania skryptu, który później wstawimy w kolejkę. Na samym początku kopiujemy przykładowy plik do naszego katalogu z obliczeniami, wraz ze zmianą nazwy:
cp /opt/examples/pbs/gaussian/g03 ~/MOLL/skrypt1
Skrypt ten ma zawartość:
#!/bin/tcsh -f
#0 G03 na halo jest smp wiec moze liczyc sie tylko na jednym nodzie:
#PBS -l nodes=1:ppn=2
#1 WPISZ czas ZEGAROWY potrzebny na wykonanie zadania: format: HH:MM:SS
#PBS -l walltime=24:00:00
#2 WPISZ ile miejsca potrzebuje twoje zadanie na dysku lokalnym /tmp1 (wazne dla MPn, CI itp) - nie wiecej niz 75GB - warto tez uzyc dyrektywy Maxdisk w inpucie Gaussiana
#PBS -l file=32gb
#ustawienia srodowiska gaussian'a: lepiej nie zmieniac!
setenv g03root /opt/gaussian
source /opt/gaussian/g03/bsd/g03.login
setenv GAUSS_SCRDIR /tmp1
#sprawdz wezel na ktorym sie liczy i jego zasoby lokalne
uname -a
df -k /tmp1
# WPISZ katalog gdzie sa inputy i gdzie beda outputy (output z gaussian)
# or checkponit. Powinien to byc podkatalog w /home
cd ~/...
date
# WPISZ zamiast test.inp i test.out nazwe swojego inputu i outputu
# Tu uruchamia sie gaussian
g03 < test.inp > test.out
date
# zakomentuj ponizsze 4 linie, jesli chcesz zachowac pliki tymczasowe
if ( `/bin/ps -ef|grep $USER|egrep "g98|g03" |grep -v "grep"|wc -l` == 0 ) then
/bin/rm -f /tmp1/Gau-*
/bin/rm -f /tmp1/*.rwf
endif
# info o zadaniu z PBSa
qstat -f $PBS_JOBID
To, co musimy zmienić, to miejsca zaznaczone kolorem czerwonym, w dalszej kolejności jak rozkręcimy się w liczeniu będziemy modyfikować bardziej skrypt. Zmieniamy:
cd ~/...
na:
cd ~/MOLL
oraz:
g03 < test.inp > test.out
na:
g03 < nazwaplikudoobliczen.inp > nazwaplikuzwynikami.out
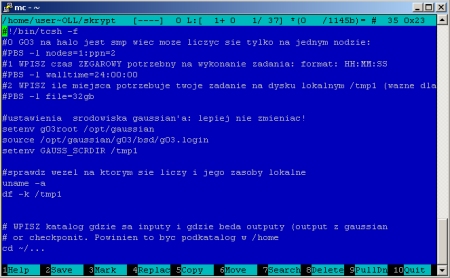
Możemy to wykonać w edytorze vi, dla osób mało obeznanych z linuxem proponuję edytor mcedit. (edytor tekstowy programu Midnight Commander). Wykonujemy polecenie:
mcedit ~/MOLL/skrypt1
Ukazuje nam się okno programu, w którym zmieniamy w sposób intuicyjny linie w pliku, a następnie zapisujemy plik wciskając F2:
Następnie wstawiamy zadanie do kolejki poleceniem:
qsub ~/MOLL/skrypt1
W katalogu MOLL powinniśmy znaleźć pliki z obliczeniami. Dla sprawdzenia czy nasze obliczenia znajdują się w kolejce używamy polecenia qstat. W wyniku czego ukazuje się lista zadań zapisanych w kolejce (Q-zadanie czeka w kolejce, R-wykonuje się). Kasujemy zadanie z kolejki poleceniem qdel i numer zadania (np.: qdel 251887):

Dla każdego pliku, który chcemy obliczyć musimy przeprowadzić poniższą procedurę nadając za każdym razem inną nazwę dla pliku skrypt1, który będzie wykonywany. Możemy oczywiście uprościć nasze wstawianie do kolejki, korzystając z własnych napisanych skryptów.
Uprzejmie proszę o przetestowanie tej procedury i policzenie jakiegoś zadania metodą AM1.
Wszelkie problemy prosimy zgłaszać w komentarzach pod tekstem.
Logowanie spod systemu Windows
Pierwszym etapem zalogowania się do komputerów znajdujących się w ICM jest uruchomienie programu, który umożliwi komunikację pomiędzy komputerami. Jednym z takich programów jest Putty. Program ten możemy pobrać za darmo pod adresem www.putty.org lub bezpośrednio korzystając z linku do putty.exe Po uruchomieniu tego programu, pojawia się nam okno:

W polu Host Name (or IP address) wpisujemy:
gw.icm.edu.pl
Rodzaj połączenia (Connection type) wybieramy SSH.
Jeżeli wykonujemy po raz pierwszy połączenie, wówczas wpisujemy dowolną nazwę, pod którą chcemy zapisać naszą sesje (Saved sessions), przykładowo może być to nazwa:
SZKOLA_MOLNET_ICM
Zapisujemy nasze ustawienie (wybieramy opcję Save). Następnym razem będziemy czytać zapisane ustawienia (opcja Load).
W nowym oknie, które otwarło się podajemy nasz login, a następnie hasło, które otrzymaliśmy ze szkoły (już wkrótce otrzyma każdy, kto podjął się projektu):
Po wpisaniu loginu oraz hasła zostaniemy proszeni o podanie nowego, dowolnie przez nas wybranego hasła.
Po zalogowaniu pojawia się okno, w którym możemy wpisywać dalsze komendy:
Komputer Delta, na który zalogowaliśmy się to tylko maszyna dostępowa, oznacza to, że poprzez nią będziemy komunikowali się z innymi maszynami ICM, na których będziemy dokonywać obliczeń.
Logowanie spod systemu Linux
Użytkownicy sytemu Linux, aby zalogować się na maszynę ICM, muszą wpisać polecenie:
ssh gw.icm.edu.pl
a następnie (podobnie jak spod systemu Windows), podajemy login, hasło oraz nasze nowe, dowolnie przez nas wybrane hasło.